
新智元报道
编辑:编辑部 HYZ
就在刚刚,AI 设计 DNA、RNA 和蛋白质序列的能力再获得颠覆性突破,研究登上 Science 封面。Evo 模型能以无与伦比的准确性,解码和设计从分子到基因组规模的对象了,合成生物学的工作方式,从此或将彻底颠覆。
Is DNA all you need?
AI 可以实现从分子到基因组尺度的预测和生成任务了!

就在刚刚,这项研究登上了 Science 封面。

来自斯坦福和 UC 伯克利的研究人员,提出了一种全新的基因组基础大模型——Evo。
利用基于深度信号处理进展的架构,Evo 扩展到了 70 亿参数,并在单核苷酸分辨率下实现了 131 千碱基的上下文长度。
目前,项目已经在 GitHub 上开源。

论文地址:https://www.science.org/doi/10.1126/science.ado9336
开源项目:https://github.com/evo-design/evo
值得一提的是,研究人员重磅发现了 DNA 的 Scaling Law!
经过 270 万个原核生物和噬菌体基因组的训练后,Evo 在 DNA、RNA 和蛋白质模态上展现出的零样本功能预测能力,可以与特定领域的语言模型相媲美,甚至直接超越。
生成合成 CRISPR-Cas 分子复合物和转座子系统的结果表明,Evo 在多模态生成任务上的表现也很出色。
此外,研究人员还首次使用语言模型,进行了蛋白质-RNA 和蛋白质-DNA 协同设计,验证了 Evo 生成的 CRISPR-Cas 分子复合物以及 IS200 和 IS605 转座子系统的功能活性。
利用从整个基因组中学习到的信息,Evo 掌握了核苷酸序列的微小变化如何影响整个生物体的适应度,并能生成长度超过 1 兆碱基的具有合理基因组架构的 DNA 序列。
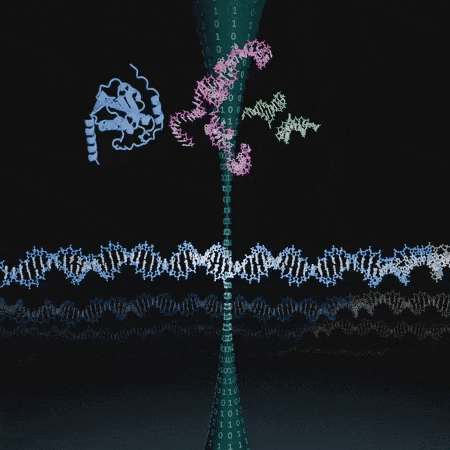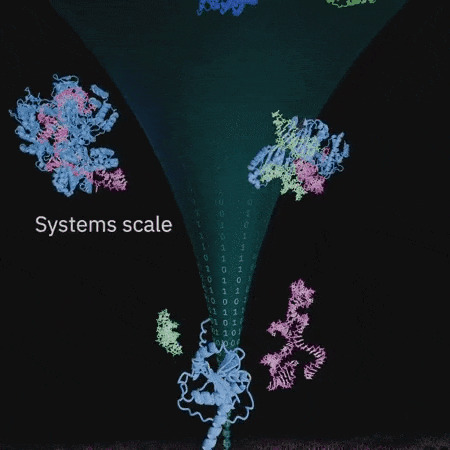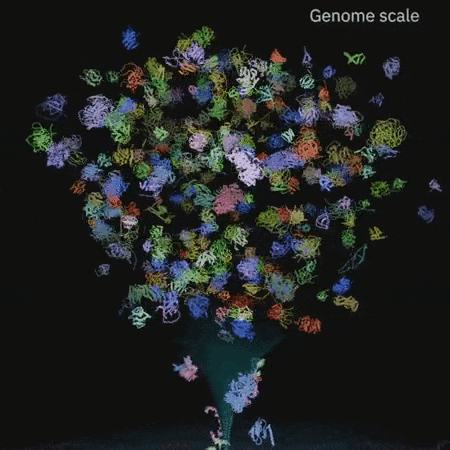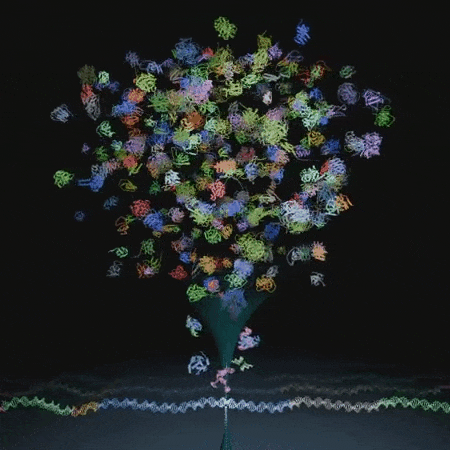
有人表示,这项研究或许能使人们逆转衰老。
世界首个 AI 生成 CRISPR-Cas 系统诞生
要知道,所有生物体的 DNA 序列中,都编码着生命的基本指令,但理解它们却很复杂。
即使是最简单的微生物基因组也是如此,数百万个碱基对,编码出 DNA、RNA 和蛋白质之间的相互作用。
这种复杂性存在于从单个分子到整个基因组的多个尺度上,代表着在进化时间中经过功能性选择的庞大遗传信息景观。
如果能有一个模型,能在保持单核苷酸分辨率的同时,还能处理大型基因组序列,就可以帮助科学家提取出自然进化变异模式中蕴含的复杂分子相互作用功能信息了。
而今 Evo 的出现,让这一切都可以实现了。
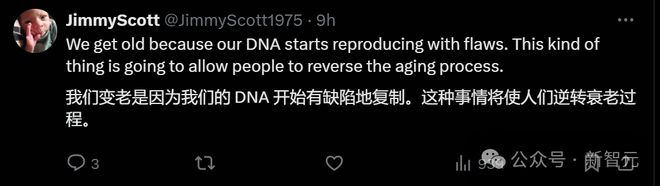
Evo 是一个包含 70 亿参数的基因组基础模型,可以学习从单个核苷酸到整个基因组的生物复杂性
它预测、生成和设计整个基因组序列的能力,可能会改变合成生物学的工作方式!
因为 Evo 了解跨模式的共同进化模式,所以研究人员决定证明它可以生成蛋白质和非编码 RNA 的大分子复合物。
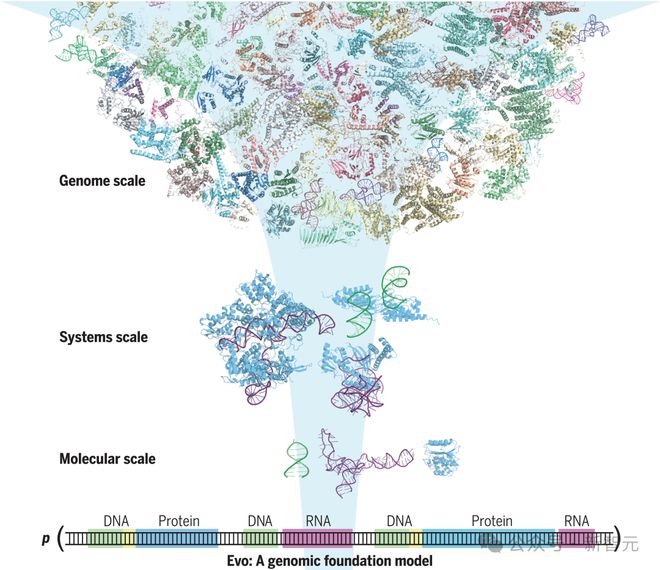
至此,世界上第一个 AI 生成的 CRISPR-Cas 系统诞生了!
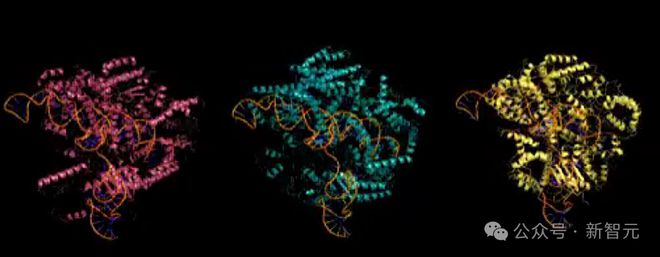
Evo 还具有生成整个基因组规模的序列的潜力。
在单个 GPU 上,研究人员生成了超过 650 KB 的 DNA 序列。使用 Evo 对这个长度的序列进行采样时可以发现,基因组包含数千个潜在的蛋白质编码序列。
未来,研究人员还将把 Evo 扩展到真核和人类序列。
研究人员表示,Evo 有极大潜力帮助或取代湿实验室实验,他对此感到非常兴奋。
很多团队都不得不对必需基因进行费力的 CRISPR 筛选,但他们直接用神经网络的前向传播将之取代了!
Evo 模型架构
如前所述,Evo 是一个基因组基础模型,共有 70 亿参数。
它通过使用单核苷酸(single-nucleotide)、字节级分词方法,在高到 131072 个 token 的上下文进行了训练。
为了有效地以核苷酸分辨率对长序列进行建模,作者利用了基于深度信号处理新兴技术的 StripedHyena 架构。
Evo 是 29 层数据控制卷积算子(hyena 层)与三层(10%)配备旋转位置嵌入(RoPE)的多头注意力交织的混合体。
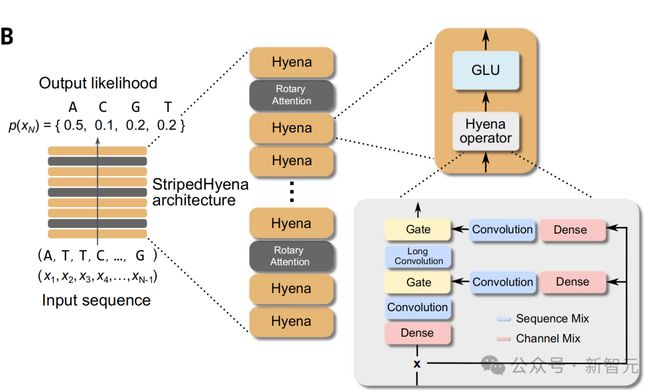
Hyena 层使用长短卷积滤波器的组合,依赖输入的方式处理序列。这使得该层在过滤 DNA 中,可能出现的噪声模式,以及将单核苷酸聚集成基序(motifs)方面特别有效。
模型混合最初是为了解决状态空间模型的缺点而提出的,最近已经证明可以提高独立 Hyena 和 Transformer 架构的语言建模的 scaling 性能。
与上一代利用 Hyena 架构的 DNA 模型 HyenaDNA 相比,Evo 基于改进的混合设计,可扩展到 1000 倍的模型大小和 100 倍的数据。
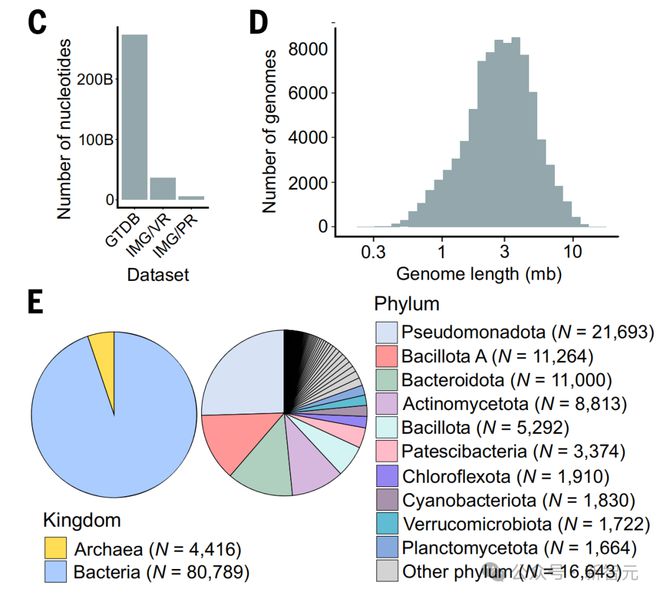
在训练模型过程中,研究人员编制了一个 OpenGenome 的大型基因组数据集,其中包含了 80000 多个细菌和古细菌基因组,以及数百万个预测的噬菌体和质粒序列,涵盖了 3000 亿个核苷酸 token。
DNA 的 Scaling Law
为了帮助 Evo 模型设计,作者对 DNA 序列建模进行了 scaling law 分析,以此确定训练、架构细节和性能指标之间的关系。
一旦获得了 scaling law,它就作为指导以最佳方式将训练 scaling 到更大的模型和数据集。
具体来说,作者在四个架构中训练了 300 多个模型:
Transformer++、Mamba、Hyena、StripedHyena。
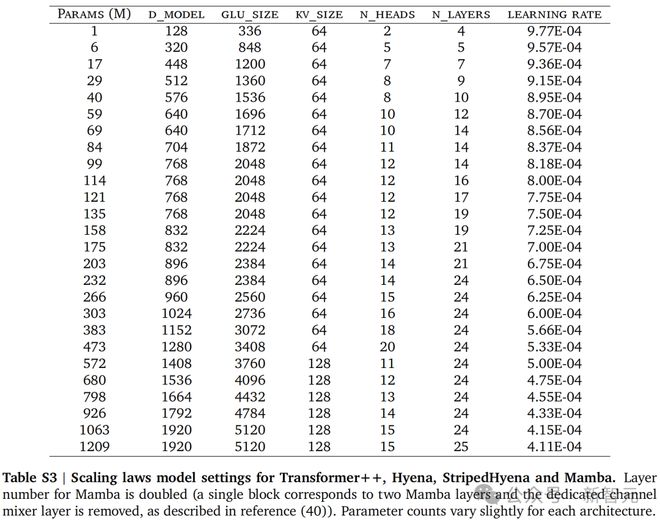
Transformer++是最先进的 Transformer,而 Mamba 是使用数据控制状态空间模型的现代架构。
结果发现,Transformer++在所有计算预算下, 产生的困惑度明显更差,字节分辨率架构效率低下的症状。
与 Transformer++ 相比,状态空间和深度信号处理架构的缩放率都有所提高,其中 Hyena 和 StripedHyena 的 scaling 率最佳。
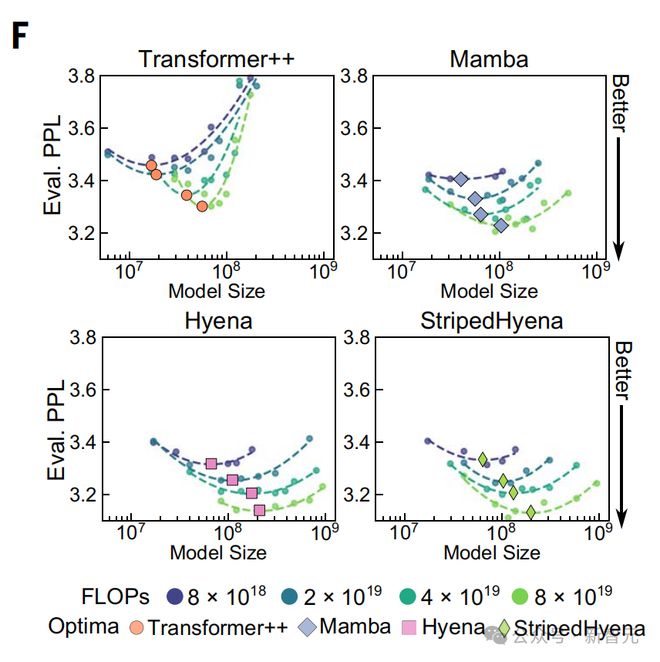
此外,在分析 sclaing 过程中,作者还观察到 StripedHyena 在所有研究的模型大小和学习率中的稳定训练。
他们还比较了架构计算最优边界之外的性能,即分配的计算预算,可能是次优的。
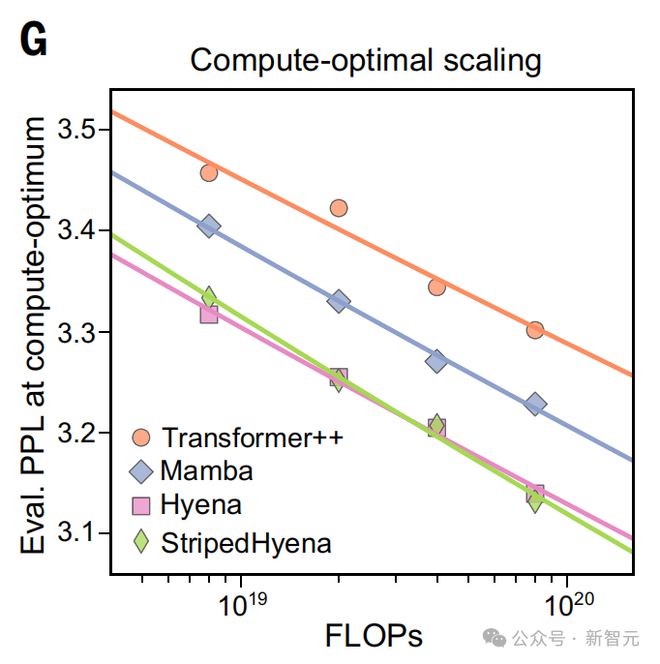
与 StripedHyena 相比,Transformer++和 Mamba 在训练过程中都经历了数值不稳定性,并且在计算最佳边界之外的 scaling 率性能下降更大。
从以上这些发现中,才使得研究人员选择 StripedHyena 作为 Evo 的架构。
Evo 跨 DNA、RNA 和蛋白质模态学习
预测突变对蛋白质功能的影响
除了评估困惑度之外,研究人员接下来研究了 Evo 在生物相关下游任务中零样本性能。
比如,在蛋白质序列或核苷酸编码序列大型语料库上,专门训练的语言模型已经证明了预测突变对蛋白质功能的影响的能力,无需任何特定任务的微调监督。
由于 Evo 的训练数据包含了蛋白质编码序列,作者测试其是否也可以进行零样本蛋白质功能预测。
这里,他们利用了深度突变扫描(DMS)研究,将一组详尽的突变引入蛋白质编码序列,然后通过实验测量这些突变对各种适应度指标的影响。
这些指标量化了功能活性。
氨基酸序列的语言模型似然或伪似然,被用来预测实验适配性得分。
为了使这项任务适用于核苷酸序列,作者使用了原始 DMS 研究中报告的野生型编码序列(wild-type coding sequence)和核苷酸突变(材料与方法)。
在原核蛋白质的 DMS 数据集上,Evo 的零样本性能超过了测试中所有其他核苷酸模型,包括 GenSLM。
Evo 还达到了与主要蛋白质特异性语言模型相媲美的性能。
先前的研究表明,对于仅使用自监督预训练的蛋白质语言模型来说,超出此性能范围的改进是困难的,这表明 Evo 已经与最先进的细菌蛋白质语言建模竞争。
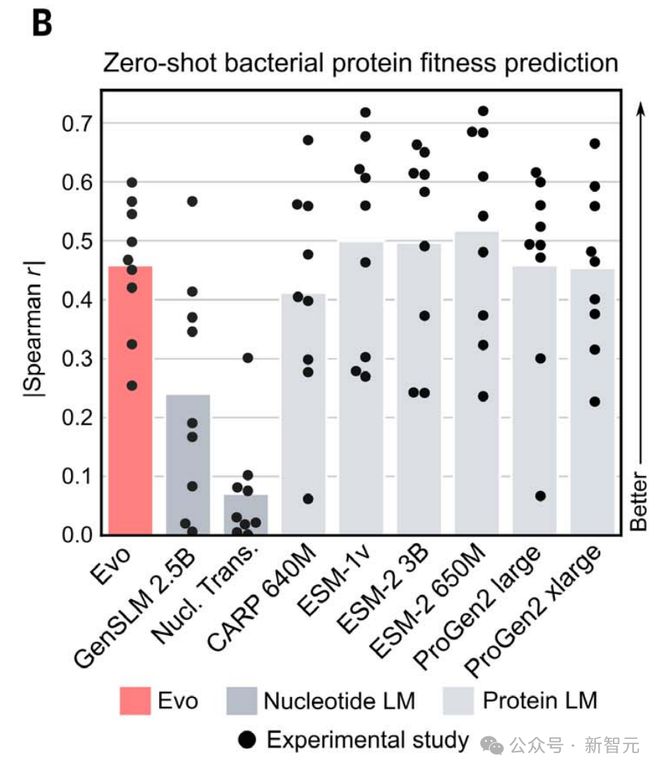
在人类蛋白质的 DMS 数据集上,Evo 无法预测突变对适应度的影响,很可能是因为预训练数据集由原核序列组成。
然而,作者还观察到野生型序列上的语言模型困惑度与适应度预测性能之间存在很强的关联性,这表明对哺乳动物编码序列进行额外的微调或未来的预训练可以提高 Evo 的性能,而不仅仅是细菌蛋白。
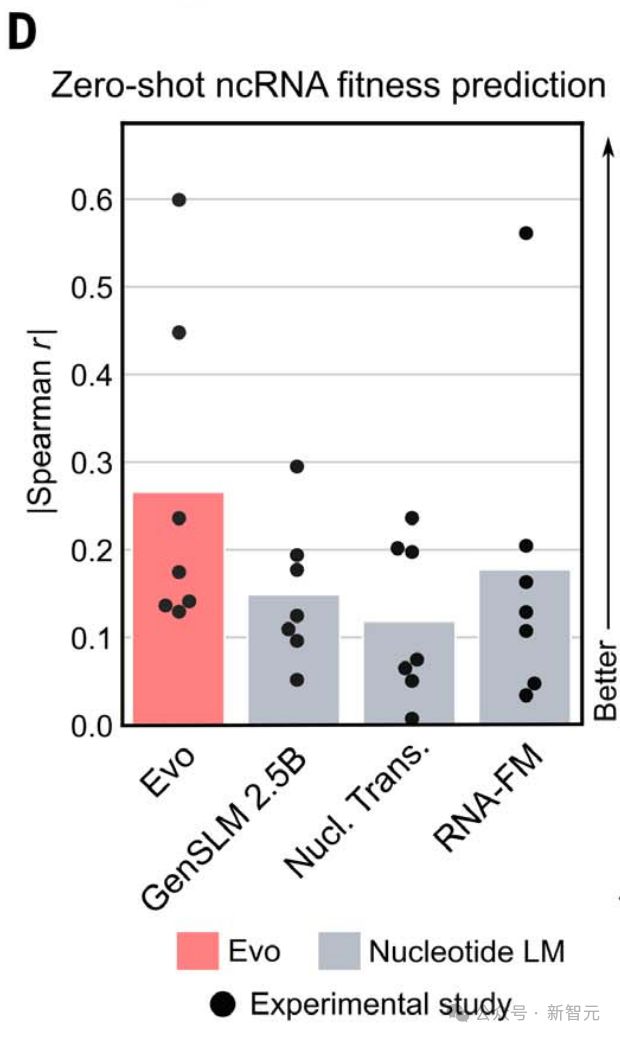
预测突变对 ncRNA 功能的影响
接下来,作者测试了相同的预训练模型是否可以学习有关的 ncRNA 功能信息,比如 tRNA、rRNA、核酶。
对此,他们收集了 ncRNA DMS 数据集并使用实验性 ncRNA DMS 研究的结果作为基础事实得分,来评估 Evo 进行零样本 ncRNA 适应性预测的能力。
结果发现,Evo 在这项任务中再次优于所有其他测试的核苷酸语言模型,包括 RNA-FM。

另外,在测量 5S rRNA 突变对大肠杆菌生长速率影响的研究中,作者观察到特别强的预测性能。
除了蛋白质序列之外,这些结果还表明 Evo 可以了解突变对 ncRNA 功能的影响。
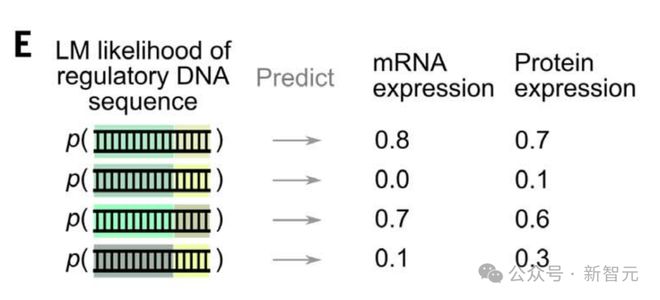
预测调控 DNA 的活性
Evo 的训练也包含了原核调控 DNA 序列,作者研究了 Evo 是否已经学习了对调控 DNA 任务的有用信息。
接下来,他们将专注于启动子序列预测基因表达和从核糖体结合位点(RBS)序列预测蛋白质表达。
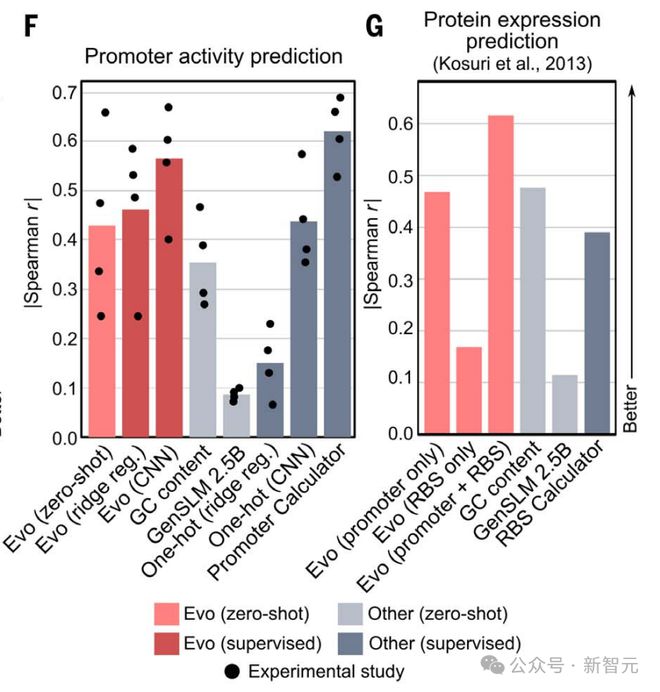
对于监督启动子活性(promoter activity)预测,作者使用来自单个研究的训练和验证分割来开发自回归模型,然后在来自其他研究的启动子数据集上测试最终模型,以评估域外泛化能力。
下图F展示了,四项研究中启动子活性与零样本语言模型可能性、序列 GC 含量或监督模型之间的相关性。
对于蛋白质表达预测,作者使用了 Kosuri 此前创建的数据集,其中除了启动子外,还包含了 RBS,除 mRNA 表达外还测量了蛋白质表达。
Evo 的 RBS 序列零样本可能性与蛋白质表达,具有弱相关性。
然而,当把启动子和 RBS 序列链接在一起时,Evo 的零样本可能性显著提高,这表明额外的调控序列,可以提供有用的功能背景。
Evo 在启动子-RBS 序列上零样本相关性,高于启动子-RBS 序列的 GC 含量、零样本 GenSLM 似然性,以及 RBS 计算器——最先进的蛋白质表达预测器。
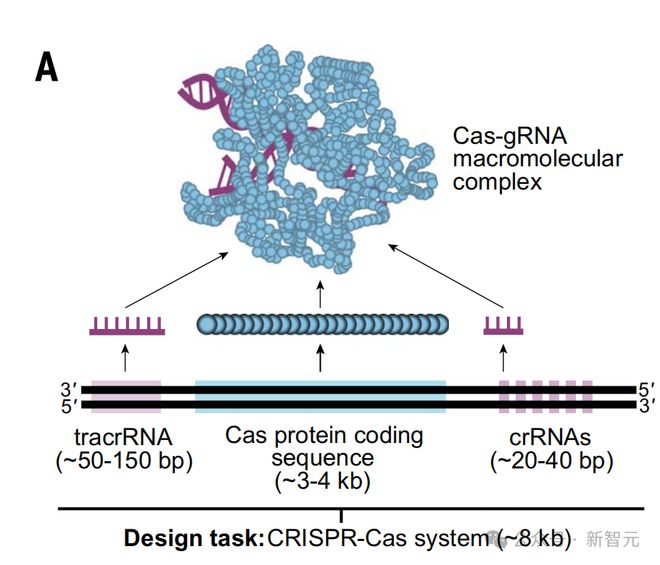
CRISPR-Cas 分子复合物的生成设计
接下来,作者推断 Evo 能够生成涉及不同分子模态之间,相互作用的功能复合物。
在原核生物中,功能相关的基因通常被组织成操纵子,并在基因组序列上彼此相邻。
因为 Evo 学习涉及上下文内任何涉及遗传元素的共变模式,所以模型应该理解编码蛋白质和 ncRNA 分子之间的相互作用。
为了证明这种能力,作者在含有 CRISPR-Cas 序列的基因组位点数据集上微调了 Evo。
值得一提的是,CRISPR-Cas 序列是由蛋白质和 ncRNA 组成的分子机器,共同引导适应性免疫对抗病毒感染。
DNA 靶向 Cas9 核酸酶,通常在 3000 到 4800 碱基对 (bp) 的编码序列中编码,并在基因组中与其同源的 CRISPR 阵列紧密相连。
CRISPR 阵列转录产生的非编码 CRISPR RNA(crRNA)分子与 Cas 蛋白结合,生成序列特异性 DNA 靶向所需的功能性防御复合物。
特别是对 Cas9 来说,第二个反式激活 CRISPR RNA(tracrRNA)与 crRNA 形成双链,从而产生一个完整的引导 RNA(gRNA)。
在细菌和古生物中发现了多种多样的 CRISPR-Cas 系统,例如基于 Cas12 或 Cas13 的系统,它们分别以 DNA 和 RNA 为靶向。
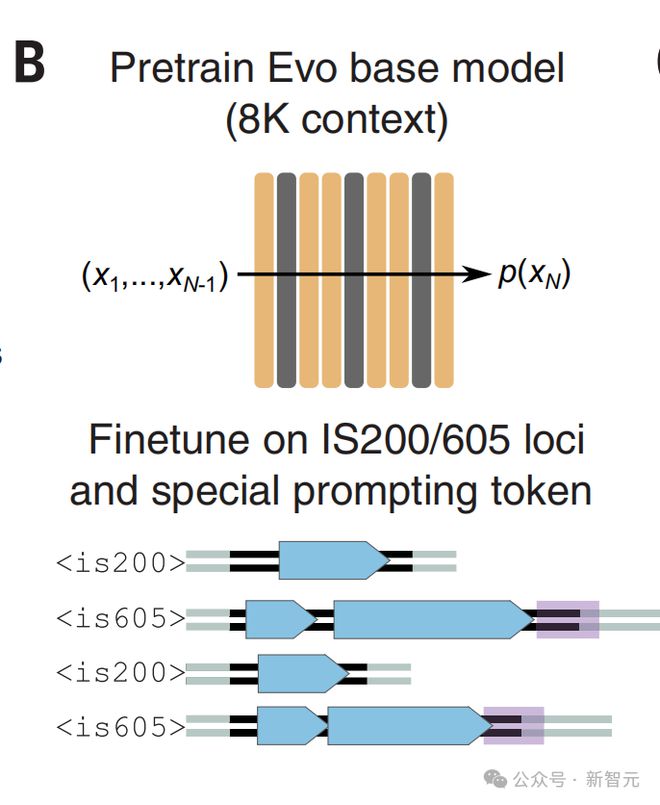
研究人员从公共宏基因组和基因组序列中提取的 72831 个 CRISPR-Cas 基因座上微调 Evo,为 Cas9,Cas 12 和 Cas 13 添加特殊的提示 token,这些标记被预先添加到每个训练序列的开头。
在采样过程中,这些 token 通过提示相应的特殊 token 知道特定 CRISPR-Cas 系统类型的生成。
使用这三种 Cas token 提示中的每一种对8-kb 序列进行采样,会产生包含 Cas 编码序列和 CRISPR 阵列的相干世代。
如果 Evo 代包含了用 MinCED 包检测的 CRISPR 阵列,以及用 Cas9、Cas 12 或 Cas 13 特征隐藏马尔科夫模型(pHMM)返回的阳性命中开放阅读框架(ORF),则将其分类为 Cas9、Cas 12 或 Cas 13 序列。
与训练数据集的序列比对显示,一些用 Cas9 pHMM 预测的 ORF 与最接近的天然 Cas9 的蛋白质序列同一性也小于 40%。
作者还发现,与仅在 CRISPR-Cas 序列上训练的模型相比,在 CRISPR-Cas 基因座上微调的 Evo 模型在所有 Cas 亚型上产生的世代质量更高、更多样化。
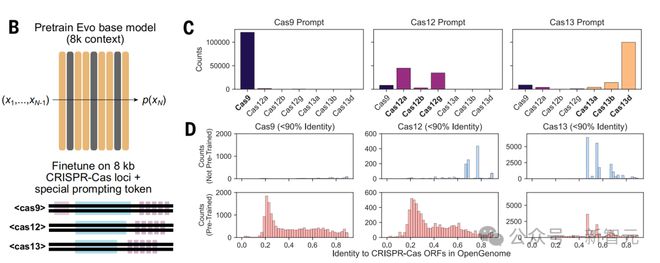
下图E展示的是,通过 pHNMR 和 CRISPR ncRNA 预测算法确定在 II 型 CRISPR 系统中,EvoCas9-1 基因中发现的核心蛋白编码基因和 ncRNA 组分。
F 是在于同源 sgRNA 和 InM DNA 靶向 10:10:1 摩尔比 Cas9:sgRNA:target 孵育后 SpCas 9 和 EvoCas 9 -1 切割反应的时程结果。
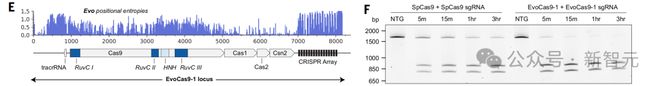
EvoCas 9 -1 氨基酸序列与用于模型微调的 Cas 蛋白数据库中,最接近的 Cas9 具有 79.9% 的同一性,与 SpCas 9 具有 73.1% 的同一性。
尽管 EvoCas 9 -1 的预测骨架结构类似于 SpCas 9 骨架结构,但 EvoCas 9 -1 的预测结构表现出更正的表面电荷分布。
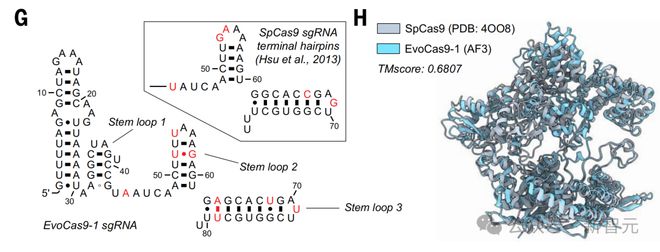
另外,来自 SpCas 9 晶体结构分离的 sgRNA 结构和通过 AlphaFold 3 模型预测的 EvoCas 9 -1 sgRNA 的结构,显示出 RNA 二级结构的强烈一致性。
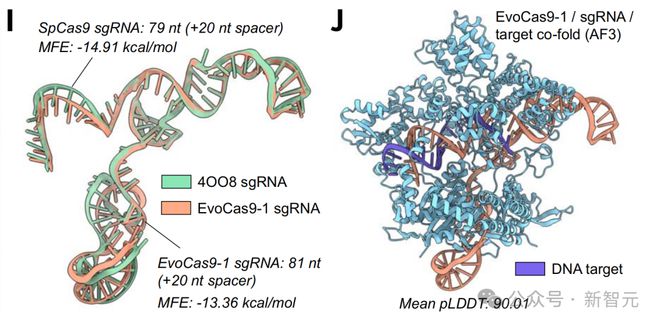
EvoCas 9 -1 的 AlphaFold 3 共折叠结构预测在其蛋白质、RNA 和 DNA 组分中,得到了平均高达 90 的 pLDDT 评分。
转座子系统的生成设计
除了分子复合物,Evo 还学习多基因系统的基本模式。
可动遗传因子(MGEs)通常包含多个基因的生物系统,并且在生命的所有领域中被发现。
它们的伺机传播推动了序列变异,新基因功能、甚至是物种的形成。
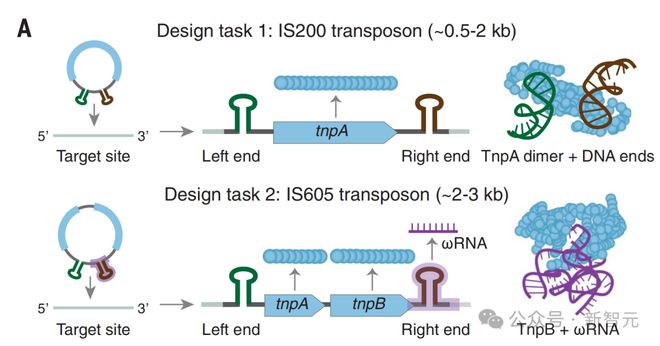
MGE 的 IS200/IS605 家族通过同源二聚体转座酶 TnpA 与元件左端和右端处的末端发夹相互作用,催化出「剥离-粘贴」转座来传播。
插入序列(IS)从单链 DNA(ssDNA)中切除,形成含有 RE-LE 结的环状产物,作为插入到新的 ssDNA 目标位点的中间产物。
IS605 元件还含有 RNA 引导的 TnpB 核酸酶和同源的ωRNA,它们偏向于转座元件的自私遗传。
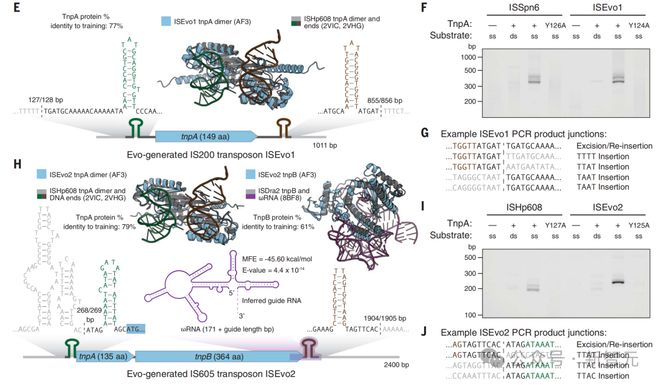
研究人员基于 10720 个 IS 605 元件和 219866 个 IS 200 元件天然序列背景下微调 Evo。
接下来,他们计算了自然 IS 200/IS 605 基因座上每个位置的条件概率的熵,并观察到熵的急剧和持续增加,特别是与元素3'端相对应,这表明了 Evo 学会了 MGE 边界的表示。
使用特殊的提示 token,研究人员使用微调模型来生成 IS200 或 IS605 元素。
在这些生成序列内检测到 TnpA 和 TnpB 蛋白质在训练集中,最接近实力的距离上变化很大,对于训练集中大于 40% 至 50% 同一性的预测结构具有一致的高 ESMFold pLDDT 值。
而且,序列长度分布与训练集中蛋白质紧密匹配。

为了选择用于实验验证的序列,作者通用与天然系统(ISSpn 6、ISStin 10、ISHp 608 和 ISDge 10)的相似性以及 TnpA 蛋白水平和 DNA 序列水平特征进行过滤,并在体外实验测试了 24 种 IS200 样和 24 种 IS605 样的设计。
然后,作者通过将体外转录产生的 TnpA 蛋白与含有假定左右端的 ssDNA 孵育,然后用外向引物进行聚合酶链反应 (PCR),以检测 TnpA 介导的切除和插入。
如果发生切除,RE-LE 结的形成会产生一条带。如果供体含有其他目标位点,并且也发生了插入,则通过相同的 PCR 反应,在两个 ssDNA 底物连接处产生条带。

研究人员观察到,24 个 Evo 生成 IS200 样元中有 11 个和 24 个 Evo 生成的 IS605 样元中,有 3 个在体外显示了切除和插入的证据。
这种活性还依赖于一个假定的催化酪氨酸的存在,以及 ssDNA 底物而不是双链 DNA(dsDNA),这与已知的 IS200/IS605 TnpA 机制一致。
为了确定每个元件的精确边界,研究人员对 PCR 产物进行了纳米孔测序。
作为对照,他们还检测了天然 IS200 元件 ISSpn6 和 IS605 元件 ISHp608,在这两种情况下,都成功地检测到了 ISFinder 标注的边界。
在生成的元件中,有三个似乎也能利用一对以上的左端或右端进行移动。含有推定 TnpB 编码序列的类 IS605 功能元件,还含有与已知ωRNAs 构建的协方差模型显著匹配(cmsearch E 值小于 0.001)的序列。
从整体上看,14 个活性元件使用了一组不同的发夹,编码的功能性 TnpA 蛋白与微调数据库的序列同一性低至 67%。
通过长基因组上下文学习基因
在第二阶段的预训练中,Evo 处理了具有 131,072 个 token 上下文的序列,其中还包含物种特异性 token。
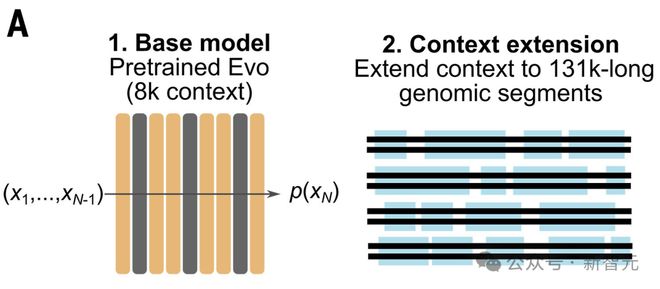
结果显示,Evo 在其 131,072 长度的上下文中,保持了单核苷酸分辨率。
这一点很重要,因为如果单个核苷酸突变破坏了该基因的表达或功能,也可能导致生命无法维持。
研究人员在给定生物体基因组中每个编码序列的开始处,插入提前终止密码子,并测量这些变化对 Evo 似然值相对于野生型序列似然值的影响。
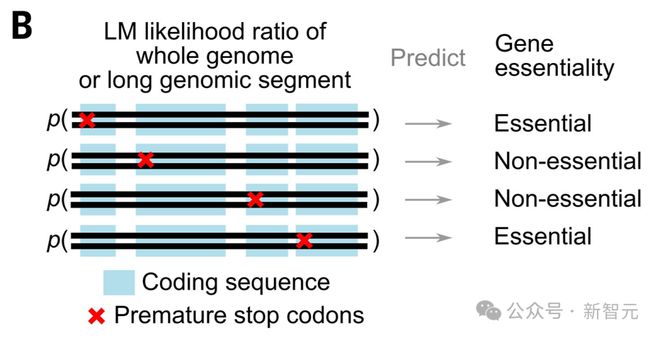
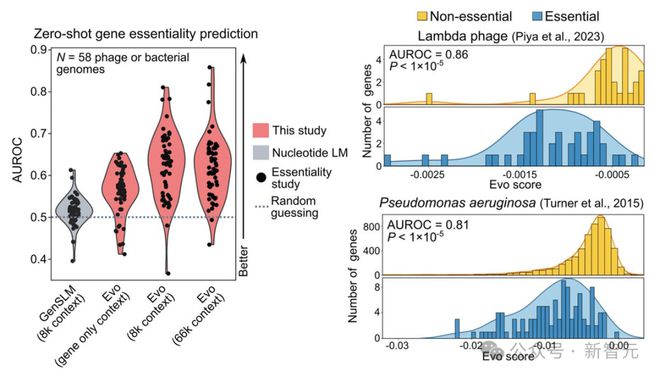
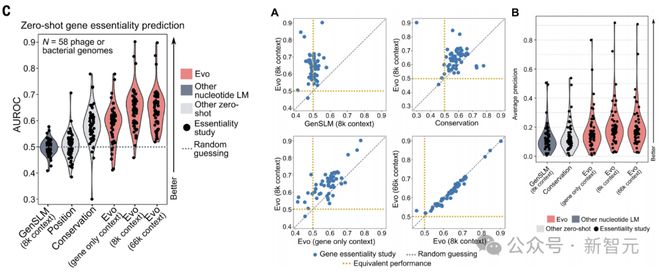
可以观察到,在 66k 上下文下,Evo 对数似然值的变化与 58 个基因组中的 49 个基因必要性显著相关。
此外还可观察到,为模型提供超出基因序列的额外基因组上下文会带来性能的显著提升,尤其是从仅基因上下文到 8k 上下文。
从 8k 到 66k 上下文,平均预测性能相当,尽管在较低范围的样本上,性能确实随着更长的上下文而提高。
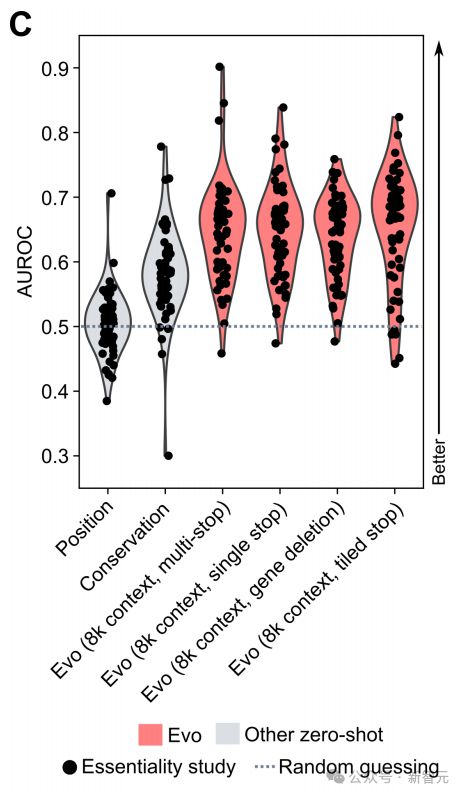
对于一些基因组,66k 上下文的 zero-shot 性能特别强,在 lambda 噬菌体必要性数据上 AUROC 达到 0.90,在铜绿假单胞菌必要性数据上 AUROC 达到 0.84。
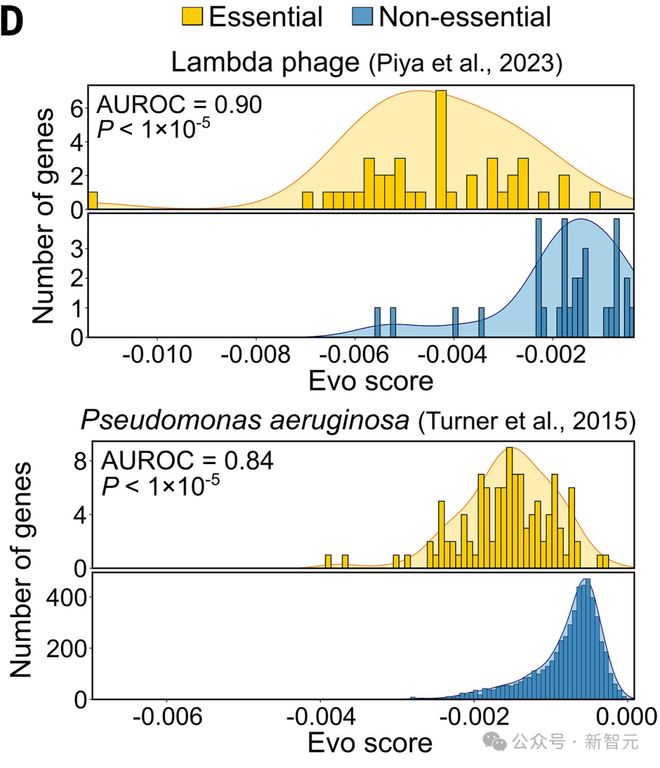
在使用不同的计算机模拟突变策略时,如改变插入终止密码子的数量或完全删除基因序列,Evo 似然值的变化也能指示基因必要性。
在基因组规模上生成 DNA 序列
研究人员使用 Evo 采样生成了 16 个各约含 1 Mb 的序列,这是模型 131 kb 上下文长度的七倍多。相比之下,「最小」的细菌基因组长度约为 580 kb。
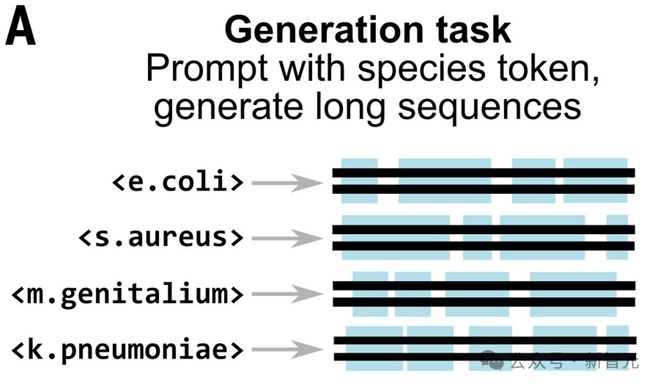
使用训练数据集中的物种级标记来提示模型生成细菌基因组
结果显示,Evo 生成的编码序列密度与自然基因组几乎相同,且明显高于随机序列。
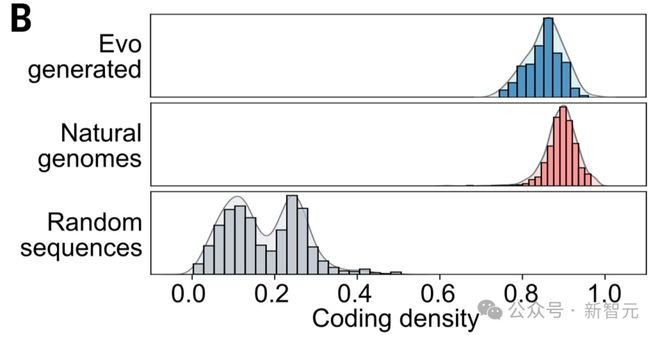
通过可视化观察,自然序列和生成序列都显示出相似的编码组织模式,邻近的序列通常具有相同的链方向;在细菌中,这些紧密相连的编码序列组通常对应于功能相关的基因簇或操纵子。

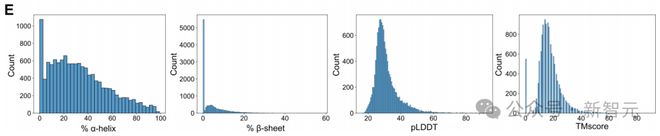
使用 ESMFold 对这些编码序列进行蛋白质结构预测时,几乎所有序列都展现出了二级结构和球状折叠。而且,很多蛋白质还展现出了与天然蛋白相似的结构。
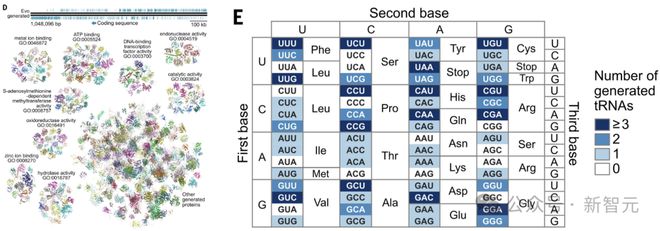
在生成的所有约 16 Mb 序列中,Evo 还能够生成 128 个 tRNA 序列,其反密码子对应于所有经典氨基酸。
进一步观察可以发现,包括 GC 含量、双核苷酸频率和某些密码子使用模式在内的各种基因组范围序列模式,与随机序列相比都更接近自然基因组。
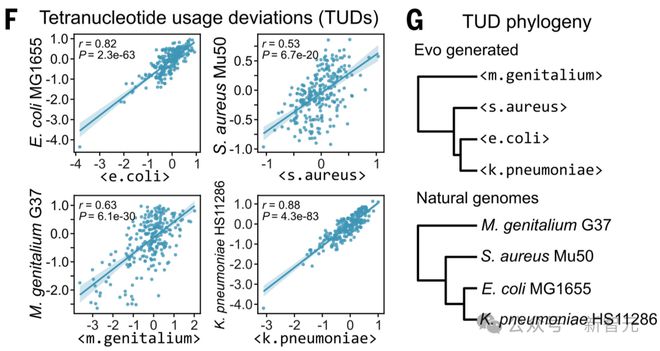
在准确性方面,Evo 的物种特异性生成序列与其对应的自然参考序列之间存在强相关性,四核苷酸使用偏差(TUDs)的准确度足以重建生成序列间的自然系统发育关系。
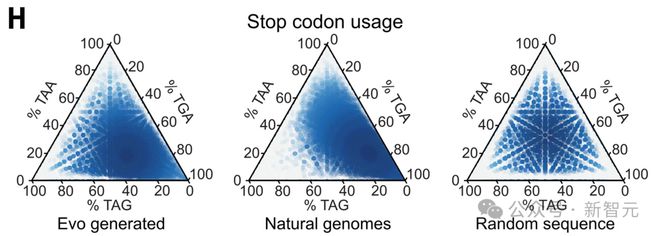
此外,TGA 和 TAA 终止密码子出现频率最高,而 TAG 最少见,这与之前在原核生物基因组中观察到的模式一致。相比之下,随机序列显示出均匀分布的终止密码子比例。
这些分析共同表明,Evo 生成的序列捕捉到了自然原核生物基因组特有的多层基因组特征。
然而,也存在一些不自然的特征。
首先,生成的序列不含有许多通常表明完整基因组的高度保守标记基因,在约 16 Mb 的样本序列中,Evo 仅生成了三个 rRNA。
其次,很多蛋白质结构预测的可信度较低,偏向于进化上较简单的α-螺旋型二级结构,且与自然蛋白质代表性数据库中的任何条目的结构匹配度有限。
目前能力有限,未来潜力无限
一个能在基因组层面设计的模型,显然有潜力推进治疗发现,拓宽我们对基础生物学的理解。

现在,球基因组与健康联盟(GA4GH)已制定了基因工程技术监管原则。
研究人员表示,已开源该模型促进透明度,同时采取措施,将真核病毒排除在了预训练数据集之外。
尽管这个第一代 DNA 基础模型能力显著,但仍有一些限制。
比如,研究人员仅仅是在 3000 亿个原核生物 token 上预训练了 Evo,仅占公开可用基因组数据中的极小部分。
另外,由于模型仅在原核生物数据上训练,在预测突变对人类蛋白质适应度的功能影响时就能力有限。
而且与自然语言模型类似,Evo 在保持长序列的连贯性和多样性方面也面临挑战。
比如许多 CRISPR-Cas 生成结果存在明显问题,如缺失或截断的 cas 基因。
在基因组层面上,虽然 Evo 生成的兆碱基长序列展示了对基因组组织的高层次理解,但在包含关键标记基因(如完整的 rRNA 集)方面仍有困难。
LLM 也遇到了相似限制,通过增加参数、标记数据、prompt 工程和人类偏好对齐一一改进,因此 DNA 模型或许也会遵循类似轨迹。
最后研究人员展望:Evo 有望成为下一代序列搜索算法的基础,将生物工程和设计的范围扩展到整个基因组的尺度。
参考资料:
https://www.science.org/doi/10.1126/science.ado9336






